DNA-Computer: Theoretisch hohe Rechenleistungen möglich
Im bislang noch relativ unerforschten Bereich sogenannter DNA-Computer ist Forschern nun ein erster konzeptioneller Schritt gelungen. Sie haben ein Design für einen DNA-Rechner entwickelt, der ihren Angaben nach ein hohes theoretisches Rechenpotenzial in bestimmten Bereichen mit sich bringt.
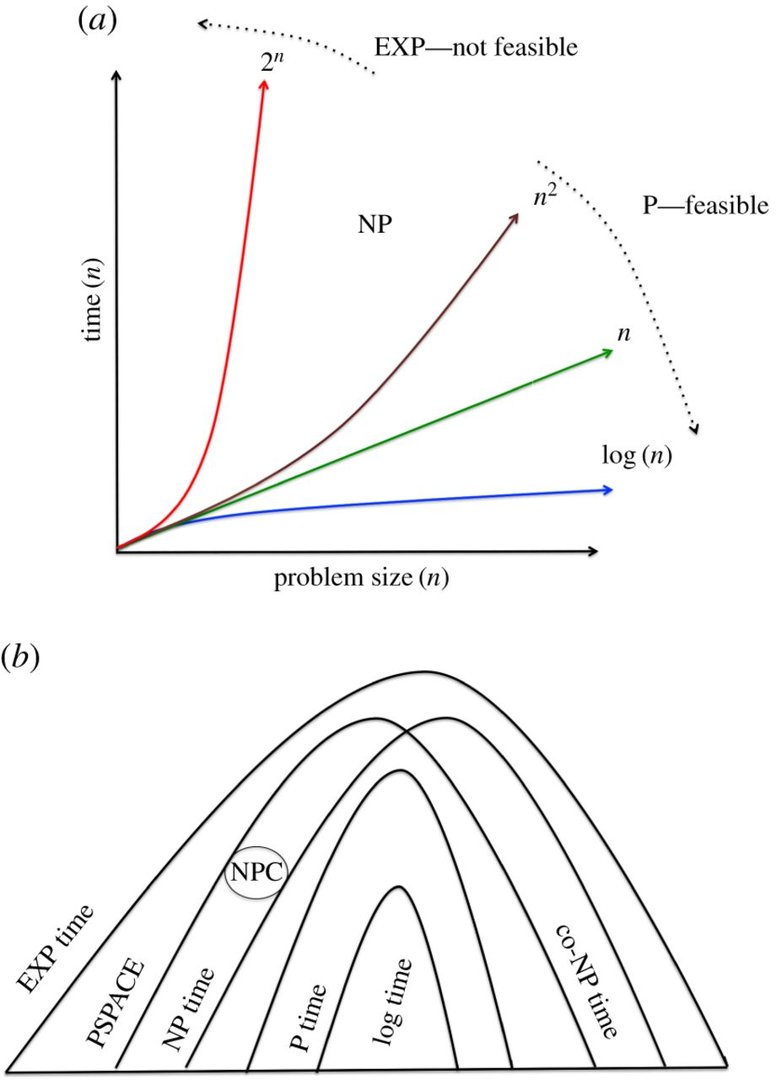
Forscher haben ein bislang eher unbeachtetes Forschungsfeld beackert und ein Konzept für die grundsätzliche Funktionsfähigkeit einer auf DNA-Nutzung basierten Recheneinheit vorgestellt. Sie haben dabei eine „Non-Deterministic-Universal-Turing-Machine“ (NUTM) entwickelt, die simpel gesagt DNA-Stränge als Prozessoren nutzt. In der Lösung bestimmter mathematischer Fragestellungen wie „non-deterministic-polynomal-complete-problems“ sollen NUTMs – zumindest theoretisch – eine exponentiell höhere Rechenleistung aufweisen als konventionelle Rechenwerke auf Siliziumbasis. Bislang wurde auf solche Versuche verzichtet, da man schon die Umsetzung für undurchführbar hielt, so zumindest die Forscher in ihrem Papier.
Universal Turing Machine
Eine Universal-Turing-Machine – benannt nach dem berühmten britischen Mathematiker Alan Turing – ist ein wesentlicher Grundpfeiler moderner Informatik. Sie ist ein ausgesprochen einfaches und mathematisch fassbares Modell der Arbeitsweise eines Computers, das wesentlich zur Entwicklung heutiger Prozessoren beitrug. Letztere stellen im weiteren Sinne eine Manifestation einer solchen Universal-Turing-Machine dar. Eine der Varianten einer solchen Maschine stellt eine NUTM oder nichtdeterministische Turingmaschine dar.
Konzeptaufbau
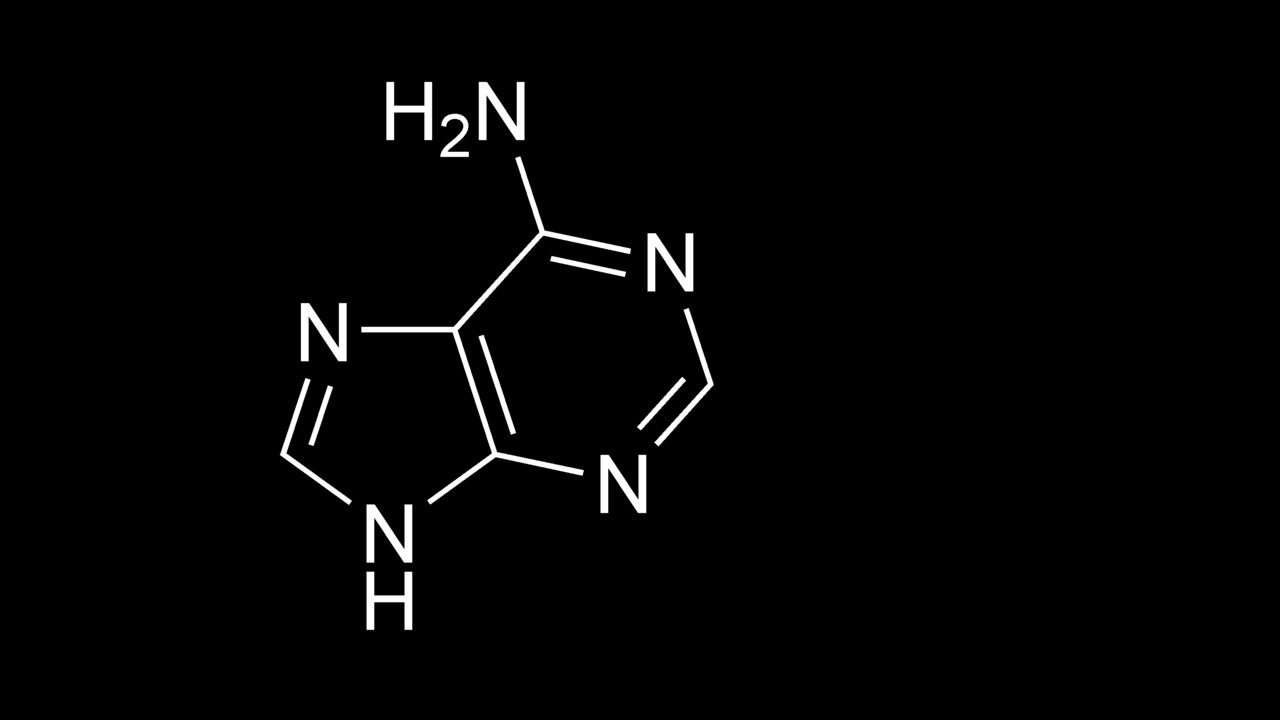
Das Design fußt auf dem sogenannten „Thue-String-Rewriting“-Ansatz. Jeder DNA-Strang stellt dabei ein encodiertes Thue-Symbol dar, das ausgelesen und modifiziert werden kann. Dieser Ansatz biete laut den Forschern Effizienzvorteile bei der Gestaltung einer NUTM. Während für UTMs eine spezifische Reihung der Berechnungen notwendig war, fällt dieses Erfordernis durch den nicht-deterministischen Charakter einer NUTM weg. Jeder „Thue-Rewriting“-Schritt wird dabei durch eine neue zweiteilige DNA-Veränderung verkörpert. Diese besteht zum Einen aus einer Polymerase-Kettenreaktion zur Applizierung von Thue-Regeln auf alle einzelnen DNA-Stränge. Zum Anderen besteht sie aus einer Mutagenese. Diese übernimmt die Aufgabe, die notwendigen Änderungen an einem DNA-Strang vorzunehmen.
-
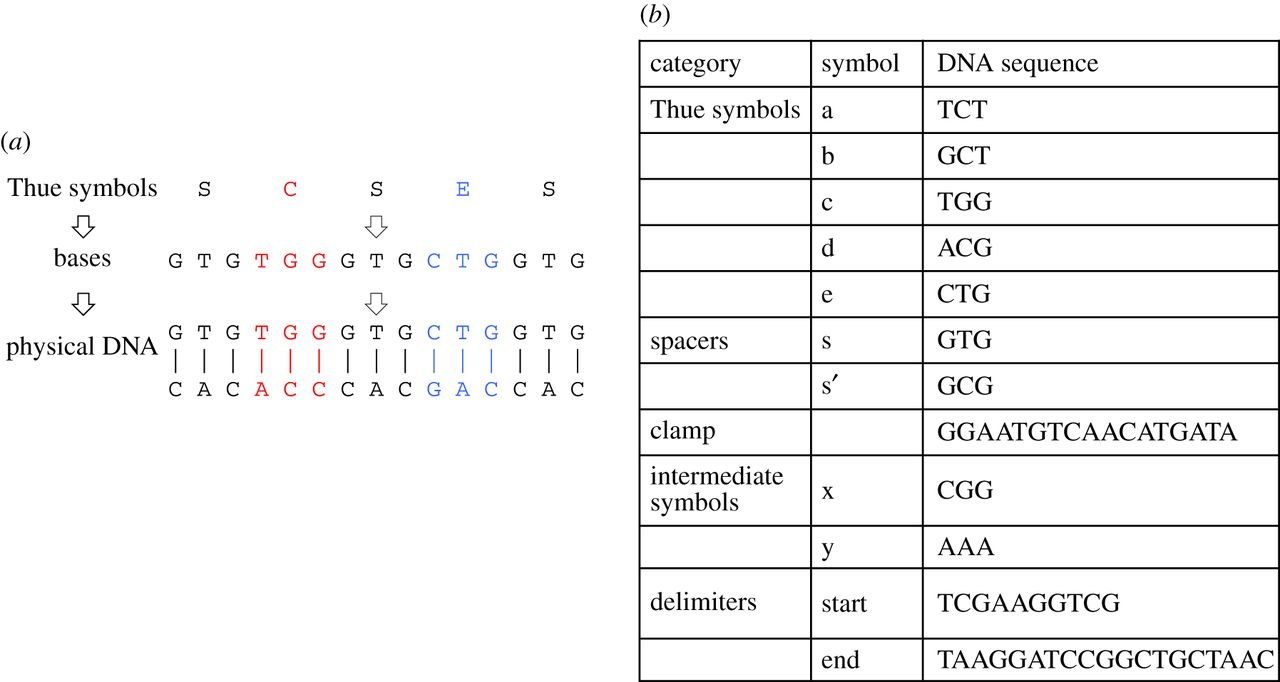 Funktionsschema eines DNA-basierten Rechenwerkes (Bild: The Royal Society)
Funktionsschema eines DNA-basierten Rechenwerkes (Bild: The Royal Society)
Vorteile ...
Die Forscher nutzen mit ihrem Design die funktionelle Fähigkeit der DNA, eine exponentielle Anzahl an Rechenwegen pro Zeiteinheit „P“ abzuarbeiten. Damit werde laut ihren Angaben – anders als bei bisherigen Rechenwerken – anstatt der Zeit der Raum für den Rechner zum limitierenden Faktor beim Lösen einer Aufgabe. Als weitere Vorteile nennen die Forscher den Umstand des sehr geringen Energieverbrauches und die Möglichkeit, per „Microprogramming“ Thue-Regeln zu encodieren. Daneben könnte man mit einer funktionierenden NUTM auch mathematischen Problemen in der Komplexitätstheorie besser zu Leibe rücken.
... und Probleme
Um eine praktische Verwendbarkeit zu erreichen, die es vermag mit moderner Computerhardware mitzuhalten, sind laut der Forschergruppe noch einige Hürden zu überwinden. Dazu zählen unter anderem eine adäquate Fehlerkorrektur, ausreichende Auslesepräzision, eine Apparatur, die die DNA-Rechenwerke beherbergen kann oder auch eine NUTM-Programmiersprache, die bis zu den Thue-Strings hin kompilierbar ist.
Die Forscher versprechen sich zudem einen möglichen Effizienzgewinn durch den zukünftigen Einsatz der CRISPR/Cas9-Methode. Diese ist im Reagenzglas aktuell noch nicht mit ausreichender Präzision einsetzbar. Auch ist die Herstellung neuer Oligonukleotide zur Adressierung der Mutationen momentan ein tagelanges Unterfangen, dass in aller Regel auf Bestellung bei darauf spezialisierten Firmen erfolgt. Diese müssen die Endprodukte dann erst wieder anliefern, bevor man sie im Labor für weiterführende Schritte einsetzen kann. Darüber hinaus haben die dann vorliegenden DNA-Stränge eine gewisse Ausschussmenge mit – durch die zelluläre DNA-Reparatur bedingte – unerwünschten Mutationen. Einer baldige Anwendbarkeit dieser Methode erscheint daher fraglich.
Zukünftige Verwendbarkeit als Prozessoren ...
Die Forscher stellen in Aussicht, dass ein irgendwann realisierbarer DNA-Computer in Desktopgröße sogar fähig sein könnte, mehr einzelne Prozessoren zu nutzen als es Mikroprozessoren auf dem Planeten gibt. Auf diese Weise sei er möglicherweise in der Lage, die schnellsten derzeitigen Superrechner leistungstechnisch auszumanövrieren und das bei einem Bruchteil der Energiekosten. Doch alleine für ein Gleichziehen mit heutiger Computerelektronik – spezifischer werden die Forscher hier nicht – würde man ein paralleles Arbeiten von 10^12 DNA-Prozessoren benötigen. Zum Vergleich sei hier angemerkt, dass gängige PCR-Thermocycler derzeit nur 96 Reaktionen zeitgleich durchführen können. Es besteht zudem zwar derzeit die grundsätzliche Möglichkeit, eine größere Menge an Polymerase-Kettenreaktionen (PCRs) gleichzeitig durchzuführen, allerdings ist die anschließende Trennung der DNA-Stränge eine komplexe Angelegenheit. Bis also all die dafür nötigen technischen Voraussetzungen geschaffen und Probleme gelöst sind, dürften sicherlich noch eine Vielzahl an Jahren ins Land ziehen.
In einem Absatz gehen die Forscher auch auf einen anderen Aspekt ihrer Arbeit ein. Ihrer Ansicht nach ist es möglich, mit ihrem NUTM-Ansatz die hochkomplexen und mehrschichtigen biochemischen Wege, über die das Verhalten einer Zelle normalerweise kontrolliert wird, zu substituieren. Man könne so die Aufgabe einer Zelle merklich einfacher neu definieren, sie sozusagen reprogrammieren. Dadurch wäre auch eine Ansteuerung über indirekte biologische Signale möglich. Man könnte so etwa ein Cytoplasma als Programm benutzen. Welches Potenzial hierin steckt, werden erst die kommenden Jahrzehnte zeigen.
... und als Speichermedium
Abseits der angedachten Verwendung von DNA-Strängen als Rechenwerke sind auch andere Nutzungsansätze im Umlauf. So hat die ETH Zürich im Februar 2015 die Langzeitstabilität von in DNA encodierten Informationen überprüft und bestätigt. Die fehlerfreie Auslese von entsprechend präparierten DNA-Datenträgern sei – abhängig von der Lagerart und Temperatur – für 2.000 Jahre bei 10 Grad Celsius und für bis zu einer Million Jahren bei minus 18 Grad Celsius möglich. Solche Zeiträume können derzeit übliche Datenträger nicht erreichen. Zeitgleich gelang es den dortigen Forschern auch, die Herstellungskosten auf etwa 500 Dollar pro Megabyte zu senken. In Anbetracht des stetig steigenden globalen Speicherbedarfes, der Prognosen zufolge zur Mitte des 21. Jahrhunderts das Angebot an Silizium überstrapazieren könnte, wäre die nicht auf Mineralvorkommen angewiesene und platzsparendere DNA-Speichermethode ein effektiver Ausweg.
Dieser Sichtweise dürfte auch Microsoft gefolgt sein, als das Unternehmen im Herbst 2015 einschlägige Tests durchführte. Bei diesen wurden Daten zuerst in DNA-Strängen gespeichert und konnten danach wieder zu 100 Prozent ausgelesen werden. Microsoft schaffte daraufhin im Frühjahr 2016 zehn Millionen künstlich hergestellter DNA-Stränge vom Unternehmen Twist Bioscience an, um intensiver eine mögliche Verwendung als Speichermedium zu erforschen. Einige Monate nach besagtem DNA-Großeinkauf ging dazu passend eine Meldung über den digitalen Äther, wonach es Microsoft gemeinsam mit der University of Washington gelungen ist, 200 Megabyte an Daten in Form von DNA-Strängen zu speichern und auch wieder auszulesen.
Ins selbe Horn stieß dann auch eine im März 2017 erschienene Arbeit von Forschern der Columbia University und dem New York Genome Center. In dieser berichteten sie, dass mittels der sogenannten DNA-Fountain-Methode in DNA-Strängen in einer Speicherdichte von 215 Petabytes pro Gramm gespeicherte Daten „perfekt“ wieder ausgelesen werden konnten. Damit würde man 85 Prozent des theoretischen DNA-Speicherlimits erreichen. Ein weiterer erfolgreicher Test erlaubte das problemlose Auslesen von Datensätzen wie unter anderem einen Film, einer größeren Textdatei und einer Kopie von KolibriOS. Einer flächendeckenden Nutzung stehen derzeit insbesondere die Kosten im Wege. Diese liegen momentan für das Synthetisieren von zwei Megabyte an Daten bei 6.650 Euro. Um die Daten wieder auslesen zu können, fallen weitere 1.900 Euro an.
